xarray-leaflet#
xarray-leaflet ist eine xarray-Erweiterung für das Plotten von gekachelten Karten. Sowohl xarray als auch Leaflet können mit Datenfragmenten arbeiten, xarray durch Dask Chunks und Leaflet durch map tiles. Mit xarray-leaflet arbeiten beide zusammen.
Installation#
Ihr könnt xarray-leaflet in eurem Jupyter-Kernel installieren mit:
$ pipenv install xarray-leaflet
Installing xarray-leaflet…
…
Standardmäßig generiert xarray-leaflet Kacheln in temporären Verzeichnissen. Bei dynamischen Karten wird bei jeder Interaktion mit der Karte ein neues Verzeichnis erstellt, entweder durch Ziehen oder Zoomen. Dies liegt daran, dass eine direkte Zuordnung zwischen dem Kachelverzeichnis und der URL besteht, unter der die Kacheln bereitgestellt werden. Da bei dynamischen Karten Kacheln nicht vom Browser zwischengespeichert werden sollten, muss sich die URL ständig ändern. Diese temporären
Verzeichnisse werden derzeit nicht automatisch bereinigt. Ihr solltet dies daher regelmäßig selbst tun. In ix-Systemen sind sie unter /tmp/xarray_leaflet_*.
Beispiel#
Um das Beispiel ausführen zu können, müsst Ihr zusätzlich die folgenden Pakete in eurem Kernel installieren:
requests
tqdm
xarray-leaflet
dask
[1]:
import os
import warnings
import zipfile
import matplotlib.pyplot as plt
import numpy as np
import requests
import rioxarray
import xarray_leaflet
from ipyleaflet import LayersControl, Map, WidgetControl, basemaps
from ipywidgets import FloatSlider
from tqdm import tqdm
Wir werden das DEM (Digital Elevation Model) für Europa aus dem HydroSHEDS-Datensatz anzeigen, das das Terrain darstellt. Laden wir zunächst die Daten herunter:
[2]:
url = 'https://edcintl.cr.usgs.gov/downloads/sciweb1/shared/hydrosheds/sa_30s_zip_grid/eu_dem_30s_grid.zip'
filename = os.path.basename(url)
name = filename[:filename.find('_grid')]
adffile = os.path.join(name, name, 'w001001.adf')
if not os.path.exists(adffile):
r = requests.get(url, stream=True)
with open(filename, 'wb') as f:
total_length = int(r.headers.get('content-length'))
for chunk in tqdm(r.iter_content(chunk_size=1024), total=(total_length/1024) + 1):
if chunk:
f.write(chunk)
f.flush()
zip = zipfile.ZipFile(filename)
zip.extractall('.')
Es ist ein Datensatz, den Rasterio öffnen kann, aber um ein schönes DataArray mit allen Metadaten zu erhalten, öffnen wir es mit rioxarray:
[3]:
da = rioxarray.open_rasterio(adffile, masked=True)
da
[3]:
<xarray.DataArray (band: 1, y: 5760, x: 9480)>
[54604800 values with dtype=float32]
Coordinates:
* band (band) int64 1
* x (x) float64 -14.0 -13.99 -13.98 -13.97 ... 64.98 64.99 65.0
* y (y) float64 60.0 59.99 59.98 59.97 ... 12.03 12.02 12.01 12.0
spatial_ref int64 0
Attributes:
scale_factor: 1.0
add_offset: 0.0Die Projektion ist EPSG:4326 (auch bekannt als WGS84). Dabei entspricht die Koordinate x den Längengraden und y den Breitengraden (in Grad). Es gibt nur ein Band.
[4]:
da = da.sel(band=1)
da.name = "DEM"
Der Datensatz kann zu groß sein, um ihn im Speicher zu halten, daher werden wir ihn in kleinere Teile zerlegen. Dies wird auch die Leistung verbessern, da die Erzeugung einer Kachel mit Dask parallel erfolgen kann.
[5]:
da = da.chunk((1000, 1000))
warnings.filterwarnings("ignore")
Wir müssen lediglich eine Karte erstellen, bevor wir sie an unsere DataArray-Erweiterung übergeben.
[6]:
m = Map(
center=[52.5162, 13.3777],
zoom=3,
basemap=basemaps.CartoDB.Positron,
interpolation="nearest",
)
m
Um unsere Daten auf der Karte darzustellen, rufen wir leaflet.plot() mit unserem DataArray auf und übergeben die Karte als Parameter. Wir erhalten eine Ebene zurück, die wir z.B. mit einem Schieberegler zur Einstellung der Deckkraft weiter steuern können.
[7]:
l = da.leaflet.plot(m, colormap=plt.cm.terrain)
l.interact(opacity=(0., 1.))
Nun fügen wir den Schieberegler in die Karte ein. Außerdem werden wir ein Ebenensteuerelement einfügen.
[8]:
layers_control = LayersControl(position="topright")
m.add_control(layers_control)
opacity_slider = FloatSlider(description="Opacity:", min=0, max=1, value=1)
def set_opacity(change):
l.opacity = change["new"]
opacity_slider.observe(set_opacity, names="value")
slider_control = WidgetControl(widget=opacity_slider, position="bottomleft")
m.add_control(slider_control)
Die select()-Methode ermöglicht es, eine Region durch Klicken und Ziehen eines Kästchens auf der Karte auszuwählen Zuvür müsst ihr jedoch auf den ⏹️-Button klicken.
[9]:
da.leaflet.select()
Ihr könnt dann das ausgewählte Datenfeld zurückerhalten und es z.B. plotten.
[10]:
da_selected = da.leaflet.get_selection()
[11]:
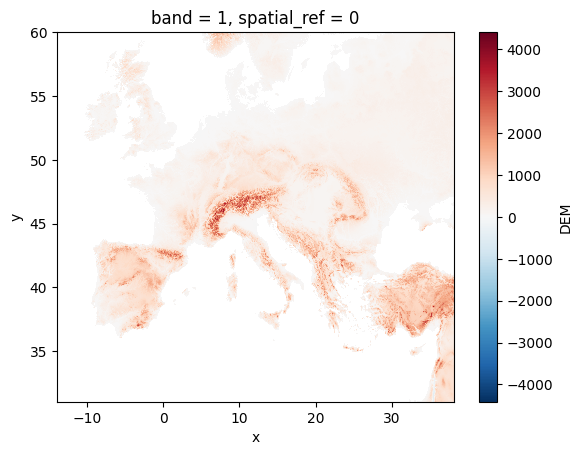
if da_selected is not None:
da_selected.plot.imshow()
Schließlich können die Auswahl und die Buttons entfernt werden:
[12]:
da.leaflet.unselect()